第一作者:王秋岩
通讯作者:王秋岩 裴晓林 谢恬
发表期刊:Communications Biology
期刊5-Year Impact Factor:5.2
通讯单位:杭州师范大学基础医学院 杭州师范大学药学院 吉林大学生科院 杭州师范大学材料与化工学院
论文DOI: https://doi.org/10.1038/s42003-025-08984-y
成果简介
近期,Communications Biology 期刊发表了杭州师范大学王秋岩、谢恬、裴晓林团队联合开展的最新研究成果,论文标题为《PAM-readID is a rapid, simple, and accurate PAM determination method for CRISPR-Cas enzymes in mammalian cells》。该研究创新性地提出了一种名为 PAM-readID 的新技术,无需依赖复杂的荧光分选步骤,仅需极低的测序深度,甚至可通过桑格测序(Sanger sequencing),就能在哺乳动物细胞中快速且精准地解析 Cas 酶的 PAM 图谱。
引言
CRISPR-Cas 核酸酶凭借简便高效特性广泛应用于基因组工程,但受原型间隔序列相邻基序(PAM)限制,且同一 Cas 酶在体外、细菌细胞、哺乳动物细胞中 PAM 谱存在固有差异。目前体外和细菌细胞的 PAM 测定方法已成熟,而哺乳动物细胞作为其核心应用场景,现有方法多依赖荧光报告系统和流式细胞分选(FACS),技术复杂、难以推广,制约了 CRISPR 技术在医学研究和基因治疗领域的应用。本研究基于 GUIDE-seq 中 dsODN 标记切割位点的思路,开发 PAM-readID 方法,旨在解决这一关键瓶颈。
图文导读
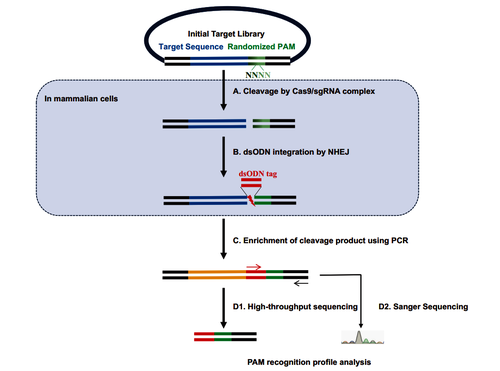
(一)PAM-readID 方法设计逻辑(图 1)
传统哺乳动物细胞 PAM 测定依赖荧光报告基因和 FACS 分选,步骤繁琐。PAM-readID 采用 “dsODN 标记 + PCR 富集” 的核心策略:构建含随机 PAM 文库的靶质粒与表达 Cas 酶和 sgRNA 的质粒,共转染哺乳动物细胞并加入 dsODN;Cas 酶识别 PAM 切割靶质粒后,NHEJ 修复会将 dsODN 整合到切割位点;再通过特异性 PCR 富集含可识别 PAM 的产物,最后经测序分析获得 PAM 谱,规避了荧光报告系统和 FACS 的依赖,简化实验流程。
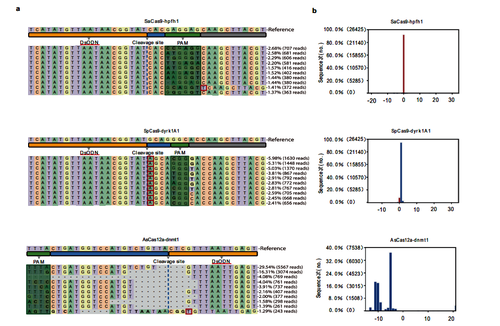
(二)dsODN 整合与 indel 特征分析(图 2)
验证显示,SaCas9 切割产物中近 99% 为单纯 dsODN 整合,SpCas9 约 90% 为 dsODN 整合结合 1bp 插入,两者 PAM 均完整;AsCas12a 因切割产生 5′ 突出末端,虽伴随 1-20bp 缺失,但仍有超 50% 序列保留完整 PAM,筛选后可用于分析,证明该方法能特异性捕获含完整 PAM 的切割产物。
(三)Cas 酶 PAM 偏好性验证(图 3)
PAM-readID 测定的 7 种 Cas 酶 PAM 谱与已知结论高度一致:SaCas9 偏好 5′-NNGRRN-3′,Nme1Cas9 偏好 5′-N4GATT-3′,SpCas9 偏好 5′-NGG-3′,AsCas12a 偏好 5′-TTTV-3′。同时成功捕获 SpG、SpRY 等突变体的 PAM 松弛特性,而这是体外方法 HT-PAMDA 难检测的。
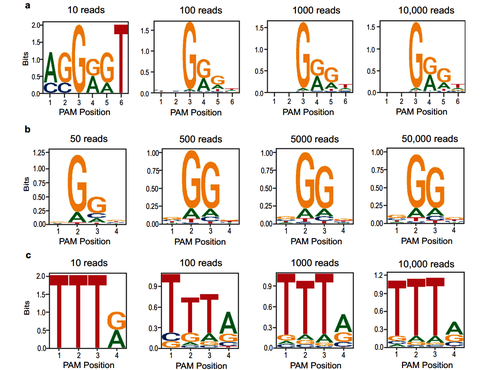
(四)测序深度与方法兼容性(图 4)
该方法测序深度需求极低:SaCas9 需 100 条读段,SpCas9 需 500 条,AsCas12a 仅需 10 条,远低于传统方法。且支持桑格测序,通过 EditR 软件分析色谱图获得的 PAM 谱与高通量测序结果一致,大幅降低技术门槛。
小结
PAM-readID 无需依赖荧光报告系统和 FACS,简化了哺乳动物细胞内 PAM 测定流程;测序深度需求低、成本低,且能准确解析多种 Cas 酶(含突变体)的 PAM 谱,包括经典与非经典 PAM。该方法为新型基因组编辑核酸酶的开发,以及基因治疗中 Cas 酶的选择与优化提供关键支撑,具有重要科研价值和应用前景。
作者简介
第一作者兼通讯作者:王秋岩,博士,副教授,硕士生导师。主要研究方向为酶法药物合成和CRISPR新酶研发,发表SCI文章20余篇,授权专利10余项。曾主持国家自然科学基金、浙江省自然科学基金、杭州市科技计划及多项企业横向课题。杭州市C类高层次人才,以主要完成人身份曾获得国家科技进步二等奖(第四完成人),教育部科技进步一等奖(第二完成人),国家发明专利金奖(第二完成人)。